RNA: Funções, Estrutura e Tipos - Ciência - 2023
science
Contente
- Estrutura
- Nucleotídeos
- Cadeia de RNA
- Forças que estabilizam o RNA
- Tipos e funções de RNA
- RNA mensageiro
- Transcrição
- Iniciação, alongamento e término
- Transcrição em procariontes
- Transcrição em eucariotos
- RNA mensageiro em eucariotos
- Splicing RNA
- RNA ribossomal
- Processamento de RNA ribossômico
- Polirribossomos
- RNA de transferência
- MicroRNA
- Silenciamento de RNA
- Diferenças entre DNA e RNA
- Origem e evolução
- Referências
o RNA ou RNA (ácido ribonucleico) é um tipo de ácido nucléico presente em organismos eucarióticos e procarióticos e em vírus. É um polímero de nucleotídeo que contém quatro tipos de bases nitrogenadas em sua estrutura: adenina, guanina, citosina e uracila.
O RNA é geralmente encontrado como uma única banda (exceto em alguns vírus), de forma linear ou em uma série de estruturas complexas. Na verdade, o RNA possui um dinamismo estrutural que não é observado na dupla hélice do DNA. Os diferentes tipos de RNA têm funções muito variadas.

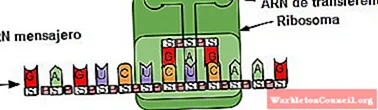
Os RNAs ribossomais fazem parte dos ribossomos, as estruturas responsáveis pela síntese de proteínas nas células. Os RNAs mensageiros funcionam como intermediários e carregam informações genéticas para o ribossomo, que traduz a mensagem de uma sequência de nucleotídeos para uma sequência de aminoácidos.
Os RNAs de transferência são responsáveis por ativar e transferir os diferentes tipos de aminoácidos -20 no total- para os ribossomos. Existe uma molécula de RNA de transferência para cada aminoácido que reconhece a sequência no RNA mensageiro.
Além disso, existem outros tipos de RNA que não estão diretamente envolvidos na síntese de proteínas e participam da regulação gênica.
Estrutura
As unidades fundamentais do RNA são os nucleotídeos. Cada nucleotídeo é composto de uma base nitrogenada (adenina, guanina, citosina e uracila), uma pentose e um grupo fosfato.
Nucleotídeos
As bases de nitrogênio são derivadas de dois compostos fundamentais: pirimidinas e purinas.
As bases derivadas de purinas são adenina e guanina e as bases derivadas de pirimidinas são citosina e uracila. Embora essas sejam as bases mais comuns, os ácidos nucléicos também podem ter outros tipos de bases que são menos comuns.
Quanto às pentoses, são unidades de d-ribose. Portanto, os nucleotídeos que compõem o RNA são chamados de “ribonucleotídeos”.
Cadeia de RNA
Os nucleotídeos estão ligados uns aos outros por ligações químicas que envolvem o grupo fosfato. Para formá-los, o grupo fosfato na extremidade 5 'de um nucleotídeo é anexado ao grupo hidroxila (-OH) na extremidade 3' do próximo nucleotídeo, criando assim uma ligação semelhante a fosfodiéster.
Ao longo da cadeia de ácido nucleico, as ligações fosfodiéster têm a mesma orientação. Portanto, há uma polaridade da fita, distinguindo entre a extremidade 3 ′ e a extremidade 5 ′.
Por convenção, a estrutura dos ácidos nucléicos é representada pela extremidade 5 'à esquerda e a extremidade 3' à direita.
O produto de RNA da transcrição do DNA é uma banda de fita simples que gira para a direita, em uma conformação helicoidal pelo empilhamento de bases. A interação entre purinas é muito maior do que a interação entre duas pirimidinas, devido ao seu tamanho.
No RNA, não se pode falar de uma estrutura secundária tradicional e de referência, como a dupla hélice do DNA. A estrutura tridimensional de cada molécula de RNA é única e complexa, comparável à das proteínas (logicamente, não podemos globalizar a estrutura das proteínas).
Forças que estabilizam o RNA
Existem interações fracas que contribuem para a estabilização do RNA, particularmente o empilhamento de bases, onde os anéis estão localizados um sobre o outro. Este fenômeno também contribui para a estabilidade da hélice do DNA.
Se a molécula de RNA encontrar uma sequência complementar, elas podem se acoplar e formar uma estrutura de fita dupla que gira para a direita. A forma predominante é o tipo A; Quanto às formas Z, só foram evidenciadas em laboratório, enquanto a forma B não foi observada.
Geralmente, existem sequências curtas (como UUGG) que estão localizadas no final do RNA e têm a particularidade de formar rotações estábulo. Essa sequência participa do dobramento da estrutura tridimensional do RNA.
Além disso, as ligações de hidrogênio podem se formar em outros locais além dos pares de bases típicos (AU e CG). Uma dessas interações ocorre entre o 2'-OH da ribose com outros grupos.
A elucidação das várias estruturas encontradas no RNA serviu para demonstrar as múltiplas funções desse ácido nucléico.
Tipos e funções de RNA
Existem duas classes de RNA: informativo e funcional. O primeiro grupo inclui RNAs que participam da síntese de proteínas e funcionam como intermediários no processo; os RNAs informativos são os RNAs mensageiros.
Em contrapartida, os RNAs pertencentes à segunda classe, os funcionais, não dão origem a uma nova molécula de proteína e o próprio RNA é o produto final. Estes são os RNAs de transferência e os RNAs ribossômicos.
Em células de mamíferos, 80% do RNA é RNA ribossomal, 15% é RNA de transferência e apenas uma pequena porção corresponde ao RNA mensageiro. Esses três tipos trabalham cooperativamente para alcançar a biossíntese de proteínas.
Existem também pequenos RNAs nucleares, pequenos RNAs citoplasmáticos e microRNAs, entre outros. Cada um dos tipos mais importantes será descrito em detalhes abaixo:
RNA mensageiro
Nos eucariotos, o DNA fica confinado ao núcleo, enquanto a síntese de proteínas ocorre no citoplasma da célula, onde se encontram os ribossomos. Devido a essa separação espacial deve haver um mediador que carregue a mensagem do núcleo para o citoplasma e essa molécula é o RNA mensageiro.
O RNA mensageiro, abreviado mRNA, é uma molécula intermediária que contém a informação codificada no DNA e que especifica uma sequência de aminoácidos que dará origem a uma proteína funcional.
O termo RNA mensageiro foi proposto em 1961 por François Jacob e Jacques Monod para descrever a porção do RNA que transmitia a mensagem do DNA aos ribossomos.
O processo de síntese de um mRNA a partir da fita de DNA é conhecido como transcrição e ocorre de forma diferenciada entre procariotos e eucariotos.
A expressão gênica é governada por vários fatores e depende das necessidades de cada célula. A transcrição é dividida em três estágios: iniciação, alongamento e término.
Transcrição
O processo de replicação do DNA, que ocorre em cada divisão celular, copia todo o cromossomo. Porém, o processo de transcrição é muito mais seletivo, lida apenas com o processamento de segmentos específicos da fita de DNA e não requer um primer.
No Escherichia coli -a bactéria mais bem estudada nas ciências da vida - a transcrição começa com o desenrolamento da dupla hélice do DNA e a alça de transcrição é formada. A enzima RNA polimerase é responsável por sintetizar RNA e, conforme a transcrição continua, a fita de DNA retorna à sua forma original.
Iniciação, alongamento e término
A transcrição não é iniciada em locais aleatórios na molécula de DNA; existem sites especializados nesse fenômeno, chamados de promotores. No E. coli A polimerase de RNA é acoplada alguns pares de bases acima da região alvo.
As sequências onde os fatores de transcrição estão acoplados são bastante conservadas entre diferentes espécies. Uma das sequências promotoras mais conhecidas é a TATA box.
No alongamento, a enzima RNA polimerase adiciona novos nucleotídeos à extremidade 3'-OH, seguindo a direção 5 'para 3'. O grupo hidroxila atua como um nucleófilo, atacando o alfa fosfato do nucleotídeo a ser adicionado. Esta reação libera um pirofosfato.
Apenas uma das fitas de DNA é usada para sintetizar o RNA mensageiro, que é copiado na direção 3 ′ para 5 ′ (a forma antiparalela da nova fita de RNA). O nucleotídeo a ser adicionado deve estar de acordo com o emparelhamento de bases: pares U com A e G com C.
A RNA polimerase interrompe o processo ao encontrar regiões ricas em citosina e guanina. Eventualmente, a nova molécula de RNA mensageiro é separada do complexo.
Transcrição em procariontes
Em procariotos, uma molécula de RNA mensageiro pode codificar para mais de uma proteína.
Quando um mRNA codifica exclusivamente para uma proteína ou polipeptídeo, é denominado mRNA monocistrônico, mas se codifica para mais de um produto proteico, o mRNA é policistrônico (observe que, neste contexto, o termo cistron se refere ao gene).
Transcrição em eucariotos
Em organismos eucarióticos, a grande maioria dos mRNAs é monocistrônica e a maquinaria de transcrição é muito mais complexa nesta linhagem de organismos. Eles são caracterizados por possuírem três RNA polimerases, denominadas I, II e III, cada uma com funções específicas.
O I é responsável por sintetizar os pré-rRNAs, o II sintetiza os RNAs mensageiros e alguns RNAs especiais. Finalmente, III cuida dos RNAs de transferência, 5S ribossômicos e outros pequenos RNAs.
RNA mensageiro em eucariotos
O RNA mensageiro sofre uma série de modificações específicas nos eucariotos. O primeiro envolve adicionar um "limite" à extremidade 5 ′. Quimicamente, o cap é um resíduo de 7-metilguanosina ligado à extremidade por uma ligação 5 ', 5'-trifosfato.
O papel desta zona é proteger o RNA da possível degradação pelas ribonucleases (enzimas que quebram o RNA em componentes menores).
Além disso, ocorre a eliminação da extremidade 3 'e são adicionados 80 a 250 resíduos de adenina. Esta estrutura é conhecida como "cauda" poliA e serve como local de ligação para várias proteínas. Quando um procarioto adquire uma cauda poliA, ele tende a estimular sua degradação.
Por outro lado, esse mensageiro é transcrito com íntrons. Os íntrons são sequências de DNA que não fazem parte do gene, mas que "interrompem" essa sequência. Os íntrons não são traduzidos e, portanto, devem ser removidos do mensageiro.
A maioria dos genes de vertebrados tem íntrons, exceto os genes que codificam para histonas. Da mesma forma, o número de íntrons em um gene pode variar de alguns a dezenas deles.

Splicing RNA
O splicing O RNA ou processo de splicing envolve a remoção de íntrons no RNA mensageiro.
Alguns íntrons encontrados em genes nucleares ou mitocondriais podem realizar o processo de emenda sem a ajuda de enzimas ou ATP. Em vez disso, o processo é realizado por reações de transesterificação. Este mecanismo foi descoberto no protozoário ciliado Tetrahymena thermophila.
Em contraste, há outro grupo de mensageiros que não são capazes de mediar seus próprios emenda, então eles precisam de maquinário adicional. Um número bastante grande de genes nucleares pertence a esse grupo.
O processo de emenda é mediado por um complexo de proteínas denominado spliceossomo ou complexo de splice. O sistema é composto de complexos de RNA especializados chamados de pequenas ribonucleoproteínas nucleares (RNPs).
Existem cinco tipos de RNP: U1, U2, U4, U5 e U6, que se encontram no núcleo e medeiam o processo de emenda.
o emenda pode produzir mais de um tipo de proteína - isso é conhecido como emenda alternativa-, uma vez que os exons são arranjados de forma diferente, criando variedades de RNAs mensageiros.

RNA ribossomal
O RNA ribossomal, abreviado como rRNA, é encontrado nos ribossomos e participa da biossíntese de proteínas. Portanto, é um componente essencial de todas as células.
O RNA ribossomal se associa a moléculas de proteína (aproximadamente 100) para dar origem a pré-subunidades ribossômicas. Eles são classificados de acordo com seu coeficiente de sedimentação, denotado pela letra S para unidades de Svedberg.
Um ribossomo é composto de duas partes: a subunidade principal e a subunidade secundária. Ambas as subunidades diferem entre procariotos e eucariotos em termos do coeficiente de sedimentação.
Os procariotos têm uma grande subunidade 50S e uma pequena subunidade 30S, enquanto nos eucariotos a subunidade grande é 60S e a pequena 40S.
Os genes que codificam para RNAs ribossômicos estão no nucléolo, uma área particular do núcleo que não é delimitada por uma membrana. Os RNAs ribossomais são transcritos nesta região pela RNA polimerase I.
Em células que sintetizam grandes quantidades de proteínas; o nucléolo é uma estrutura proeminente. No entanto, quando a célula em questão não requer um grande número de produtos proteicos, o nucléolo é uma estrutura quase imperceptível.
Processamento de RNA ribossômico
A grande subunidade ribossomal 60S está associada aos fragmentos 28S e 5.8S. Em relação à subunidade pequena (40S), ela está associada ao 18S.
Em eucariotos superiores, o pré-rRNA é codificado em uma unidade de transcrição 45S, que envolve a RNA polimerase I.Esta transcrição é processada em RNAs ribossômicos 28S, 18S e 5.8S maduros.
Conforme a síntese continua, o pré-rRNA se associa a diferentes proteínas e forma partículas de ribonucleoproteína. Este sofre uma série de modificações subsequentes, incluindo a metilação do grupo ribose 2'-OH e a conversão de resíduos de uridina em pseudouridina.
A região onde essas mudanças ocorrerão é controlada por mais de 150 pequenas moléculas de RNA nucleolar, que têm a capacidade de se acoplar ao pré-rRNA.
Ao contrário do resto dos pré-rRNAs, o 5S é transcrito pela RNA polimerase III no nucleoplasma e não dentro do nucléolo. Depois de sintetizado, ele é levado ao nucléolo para se reunir com o 28S e o 5.8S, formando assim as unidades ribossomais.
Ao final do processo de montagem, as subunidades são transferidas para o citoplasma através dos poros nucleares.
Polirribossomos
Pode acontecer que uma molécula de RNA mensageiro dê origem a várias proteínas ao mesmo tempo, ligando-se a mais de um ribossomo. À medida que o processo de tradução avança, a ponta do mensageiro fica livre e pode ser ocupada por outro ribossomo, iniciando uma nova síntese.
Portanto, é comum encontrar ribossomos agrupados (entre 3 e 10) em uma única molécula de RNA mensageiro, sendo esse grupo denominado polirribossomo.
RNA de transferência
O RNA de transferência é responsável pela transferência de aminoácidos à medida que o processo de síntese de proteínas avança. Eles são compostos de aproximadamente 80 nucleotídeos (em comparação com o RNA mensageiro, é uma molécula "pequena").
A estrutura tem dobras e cruzes que lembram um trevo de três braços. Um anel adenílico está localizado em uma das extremidades, onde o grupo hidroxila da ribose medeia a união com o aminoácido a ser transportado.
Os diferentes RNAs de transferência combinam-se exclusivamente com um dos vinte aminoácidos que constituem as proteínas; em outras palavras, é o veículo que carrega os blocos de construção das proteínas. O complexo do RNA de transferência junto com o aminoácido é denominado aminoacil-tRNA.
Além disso, no processo de tradução - que ocorre graças aos ribossomos - cada RNA de transferência reconhece um códon específico no RNA mensageiro. Ao reconhecê-lo, o aminoácido correspondente é liberado e passa a fazer parte do peptídeo sintetizado.
Para reconhecer o tipo de aminoácido que deve ser entregue, o RNA tem um “anticódon” localizado na região intermediária da molécula. Este anticódon é capaz de formar ligações de hidrogênio com bases complementares presentes no DNA mensageiro.

MicroRNA
MicroRNAs ou miRNAs são um tipo de RNA curto, de fita simples, entre 21 e 23 nucleotídeos, cuja função é regular a expressão de genes. Como não é traduzido em proteína, costuma ser chamado de RNA antisense.
Como os outros tipos de RNA, o processamento de microRNA é complexo e envolve várias proteínas.
MicroRNAs surgem de precursores mais longos chamados mi-priRNA, derivados do primeiro transcrito do gene. No núcleo da célula, esses precursores são modificados no complexo do microprocessador e o resultado é um pré-miRNA.
Pré-miRNAs são grampos de cabelo de 70 nucleotídeos que continuam a ser processados no citoplasma por uma enzima chamada Dicer, que monta o complexo de silenciamento induzido por RNA (RISC) e, finalmente, o miRNA é sintetizado.
Esses RNAs são capazes de regular a expressão de genes, uma vez que são complementares a RNAs mensageiros específicos. Ao acasalar com seu alvo, os miRNAs são capazes de suprimir o mensageiro ou mesmo degradá-lo. Consequentemente, o ribossomo não pode traduzir a referida transcrição.

Silenciamento de RNA
Um tipo particular de microRNA são pequenos RNAs de interferência (siRNAs), também chamados de RNAs de silenciamento. São RNAs curtos, entre 20 a 25 nucleotídeos, que dificultam a expressão de certos genes.
São instrumentos de pesquisa muito promissores, pois permitem silenciar um gene de interesse e assim estudar sua possível função.
Diferenças entre DNA e RNA
Embora o DNA e o RNA sejam ácidos nucléicos e possam parecer muito semelhantes à primeira vista, eles diferem em várias de suas propriedades químicas e estruturais. O DNA é uma molécula de banda dupla, enquanto o RNA é de banda única.
Como tal, o RNA é uma molécula mais versátil e pode assumir uma ampla variedade de formas tridimensionais. No entanto, certos vírus possuem RNA de banda dupla em seu material genético.
Nos nucleotídeos do RNA, a molécula de açúcar é uma ribose, enquanto no DNA é uma desoxirribose, diferindo apenas na presença de um átomo de oxigênio.
A ligação fosfodiéster na espinha dorsal do DNA e do RNA está sujeita a um lento processo de hidrólise sem a presença de enzimas. Em condições alcalinas, o RNA hidrolisa rapidamente - graças ao grupo hidroxila extra - enquanto o DNA não.
Da mesma forma, as bases nitrogenadas que constituem os nucleotídeos do DNA são guanina, adenina, timina e citosina; em contraste, no RNA, a timina é substituída por uracila. O uracil pode emparelhar com a adenina, da mesma forma que a timina no DNA.

Origem e evolução
O RNA é a única molécula conhecida capaz de armazenar informações e catalisar reações químicas ao mesmo tempo; Por esse motivo, vários autores propõem que a molécula de RNA foi crucial na origem da vida. Surpreendentemente, os substratos dos ribossomos são outras moléculas de RNA.
A descoberta das ribozimas levou à redefinição bioquímica de "enzima" - como anteriormente o termo era usado exclusivamente para proteínas com atividade catalítica -, e ajudou a sustentar um cenário onde as primeiras formas de vida utilizavam apenas RNA como material genético.
Referências
- Alberts B, Johnson A, Lewis J, et al. (2002). Biologia molecular da célula. 4ª edição. Nova York: Garland Science. Do DNA ao RNA. Disponível em: ncbi.nlm.nih.gov
- Berg, J. M., Stryer, L., & Tymoczko, J. L. (2007). Bioquímica. Eu inverti.
- Campbell, N. A., & Reece, J. B. (2007). biologia. Panamerican Medical Ed.
- Griffiths, A.J.F., Gelbart, W.M., Miller, J.H., et al. (1999). Análise Genética Moderna. Nova York: W. H. Freeman. Genes e RNA. Disponível em: ncbi.nlm.nih.gov
- Guyton, A. C., Hall, J. E., & Guyton, A. C. (2006). Tratado de fisiologia médica. Elsevier.
- Hall, J. E. (2015). Livro didático de fisiologia médica de Guyton e Hall e - Livro. Elsevier Health Sciences.
- Lodish, H., Berk, A., Zipursky, S.L., et al. (2000) Molecular Cell Biology. 4ª edição. Nova York: W. H. Freeman. Seção 11.6, Processamento de rRNA e tRNA. Disponível em: ncbi.nlm.nih.gov
- Nelson, D. L., Lehninger, A. L., & Cox, M. M. (2008). Princípios de bioquímica de Lehninger. Macmillan.


